
碱基编辑技术(Base editing)是基于CRISPR/Cas系统发展起来的新型靶基因修饰技术,在2017年被Science杂志评为年度十大科学技术突破之一。现有碱基编辑技术可编辑的类型局限于C:G>T:A或A:T>G:C的碱基替换。虽然利用同源重组等方法理论上也可以实现任意类型碱基的编辑,但因其极低的效率和复杂的操作,应用范围也相对有限。2019年10月,哈佛大学DavidLiu研究团队在哺乳动物中开发了基因组引导编辑系统,为在植物中进行碱基自由编辑提供了新的方向和思路。
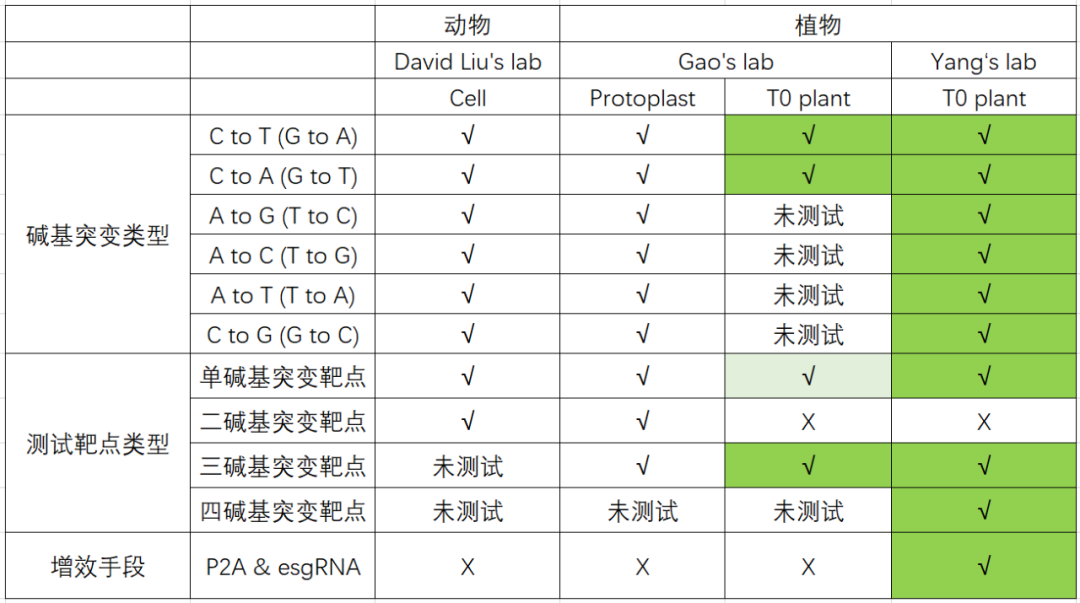
近日,北京市农林科学院玉米DNA指纹及分子育种北京市重点实验室基因组编辑团队在Cell旗下植物顶级期刊Molecular Plant(即时IF=13.1,生物学Q1期刊)上发表了题为“Versatile Nucleotides Substitution in Plant Using an Improved Prime Editing System”的研究文章,成功建立了一种高效的植物引导编辑技术,突破了原来碱基编辑技术的限制,实现了碱基转换(C:G>T:A和A:T>G:C)及碱基颠换(C:G>G(A):C(T)和A:T>T(C):A(G)),尤其在面对多碱基编辑情况时,该技术更加高效和精准。
本研究首先对动物细胞中报道的引导编辑技术系统进行了启动子置换和密码子优化,并在稳定转化的T0苗中进行了技术测试,结果发现该系统虽然工作,但与在动物细胞中相比,效率普遍较低。当利用自切割多肽串联表达PE系统和筛选标记,改由玉米的泛素基因启动子驱动,并配合使用强化sgRNA引导时,引导编辑技术的效率得以大幅提升,平均提升8倍以上,其中增效最高的靶点效率提升至26%,增加了21倍,而且在原来不工作的绝大多数靶点上也都实现了碱基的精确编辑。就精准性而言,除了在两个效率较高的靶点上检测到了少量非预期插入和删除外,其他靶点都仅含有预先设计的碱基替代,表明该技术的精确度是可以信赖的。
值得一提的是,本研究选择的目标靶点大部分都是在生产实践中有重要应用价值,但现有碱基编辑技术不能编辑或编辑效率极低的靶点,如吡氟氯禾灵除草剂抗性位点和乙酰乳酸合成酶抑制剂抗性位点等,引导编辑技术在这些位点上的编辑成功,显示了该技术在作物精准快速育种方面的潜力。
本研究与最近在Nature biotechnology上及同期背对背发表的相关研究都是聚焦于植物引导编辑技术,有些数据可以相互验证,但大家在基础系统及优化手段上各不相同,并在增效办法上具有很强的互补性,共同为未来的植物引导编辑技术优化和应用提供了基础,一起为植物功能基因组学优异等位基因研究、水稻、玉米等重要农作物的快速、精准育种提供技术支撑。
徐雯、张成伟为本文共同第一作者,杨进孝为通讯作者。本研究得到北京学者(BSP041)和北京市农林科学院创新团队项目(JNKYT201603)资助。
DOI:https://doi.org/10.1016/j.molp.2020.03.012